
近日,姜雨教授团队在基因组结构变异(Structural Variations,SVs)准确分型的研究领域取得重要突破。最新成果以《SVLearn: a dual-reference machine learning approach enables accurate cross-species genotyping of structural variants》为题,在国际期刊Nature Communications上在线发表。学院博士研究生杨启蒙、牛津大学博士后孙鉴锋为该论文共同第一作者,我校姜雨教授和蔡钰东博士后为共同通讯作者。我校信息学院刘全中副教授,德国亥姆霍兹研究中心汝锦龙博士后参与相关研究工作。
姜雨教授团队长期致力于基因组结构变异(Structural variations,SVs)相关研究。曾开发了可在动植物大群体中快速检测基因组拷贝数变异的软件CNVcaller。近年来,随着越来越多高质量结构变异数据集的产生,大规模群体研究普遍采用短读长测序数据进行结构变异基因分型。然而,受限于短读长和参考序列的偏差,结构变异的分型准确度仍然面临挑战。
为有效解决这一问题,团队创新性地提出了基于双参考基因组的机器学习策略,开发出SVLearn软件。该工具通过同时使用原始参考基因组和包含变异序列的替代基因组,全面整合了基因组特征、比对特征和基因型特征,分别在人、牛、绵羊数据集合中建立了高精度的结构变异机器学习分型模型。
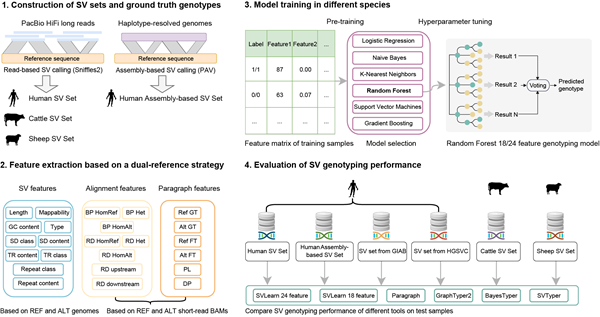
研究团队利用三个物种共六个高质量结构变异数据集,对SVLearn与四种国际主流工具进行了全面的性能评估。结果显示,SVLearn整体性能表现有5%-10%以上的提升,将最终分型准确率提升到90%左右,在复杂的基因组重复区域,结构变异的分型准确性提升尤为显著。值得注意的是,针对测序深度较低的数据,团队专门进行了模型优化,使得SVLearn在低覆盖度下依然能保持与30倍测序深度相当的分型效果,大大提高了大规模结构变异研究的经济性和可行性。此外,通过跨物种交叉验证,进一步证实了该工具的普适性,表明其在不同物种中具有较强的基因分型能力。这一创新方法的提出,为结构变异研究提供了全新的分析工具,并有望推动人类疾病研究、动植物育种等领域的快速发展。
本研究得到国家重点研发计划(2023YFD1300402,2022YFF1000100)、国家自然科学基金(U21A20120)、陕西省畜禽育种双链融合重点项目(2022GD-TSLD-46-0401)的资助。感谢西北农林科技大学高性能计算平台提供计算资源。
编辑:张晴
终审:刘玉峰

西北农林科技大学

西农深圳研究院
